Veamos un ejemplo en el que PCA (o LDA) no serían de utilidad: Scikit-Learn incluye la clase make_moons que permite generar puntos aleatorios con forma de dos lunas, puntos pertenecientes, por defecto, a dos clases::
[ 1.71947411, -0.19596548],
[ 0.44507816, -0.23873905],
[ 1.51863492, -0.39084982],
[ 0.34573656, -0.21692838]])
Mostremos estos datos gráficamente:

En este escenario, extraer el componente principal del dataset sería muy poco útil:
X_pca = pca.fit_transform(X)
Mostremos los primeros cinco valores del resultado:
[-1.26618544],
[-0.50553939],
[ 0.40741947],
[-0.73634878]])
Si queremos mostrarlos gráficamente, nuestra función show_boundaries requiere una estructura de dos dimensiones, de forma que añadamos una segunda columna con ceros:
X_pca.reshape(-1, 1),
np.zeros(shape = (X_pca.shape[0], 1))
], axis = 1)
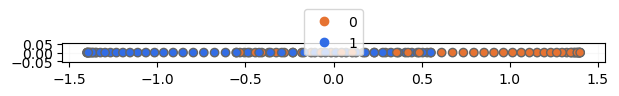
Ahora ya podemos visualizar los datos:
Los datos resultantes de aplicar PCA no permiten separar linealmente las dos clases.